主成分分析(PCA)
主成分分析(Principal Component Analysis)是目前为止最流行的降维算法。首先它找到接近数据集分布的超平面,然后将所有的数据都投影到这个超平面上。
保留(最大)方差
在将训练集投影到较低维超平面之前,您首先需要选择正确的超平面。例如图 8-7 左侧是一个简单的二维数据集,以及三个不同的轴(即一维超平面)。图右边是将数据集投影到每个轴上的结果。正如你所看到的,投影到实线上保留了最大方差,而在点线上的投影只保留了非常小的方差,投影到虚线上保留的方差则处于上述两者之间。
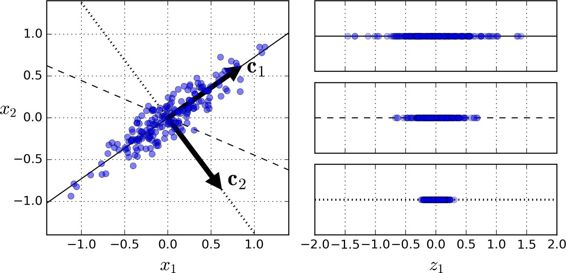
图 8-7 选择投射到哪一个子空间
选择保持最大方差的轴看起来是合理的,因为它很可能比其他投影损失更少的信息。证明这种选择的另一种方法是,选择这个轴使得将原始数据集投影到该轴上的均方距离最小。这是就 PCA 背后的思想,相当简单。
主成分(Principle Componets)
PCA 寻找训练集中可获得最大方差的轴。在图 8-7 中,它是一条实线。它还发现了一个与第一个轴正交的第二个轴,选择它可以获得最大的残差。在这个 2D 例子中,没有选择:就只有这条点线。但如果在一个更高维的数据集中,PCA 也可以找到与前两个轴正交的第三个轴,以及与数据集中维数相同的第四个轴,第五个轴等。
定义第i个轴的单位矢量被称为第i个主成分(PC)。在图 8-7 中,第一个 PC 是c1,第二个 PC 是c2。在图 8-2 中,前两个 PC 用平面中的正交箭头表示,第三个 PC 与上述 PC 形成的平面正交(指向上或下)。
概述: 主成分的方向不稳定:如果您稍微打乱一下训练集并再次运行 PCA,则某些新 PC 可能会指向与原始 PC 方向相反。但是,它们通常仍位于同一轴线上。在某些情况下,一对 PC 甚至可能会旋转或交换,但它们定义的平面通常保持不变。
那么如何找到训练集的主成分呢?幸运的是,有一种称为奇异值分解(SVD)的标准矩阵分解技术,可以将训练集矩阵X分解为三个矩阵U·Σ·V^T的点积,其中V^T包含我们想要的所有主成分,如公式 8-1 所示。
公式 8-1 主成分矩阵
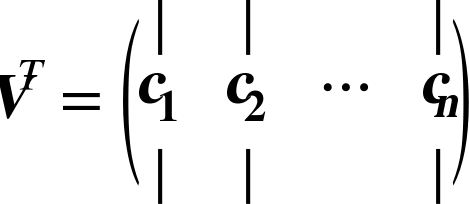
下面的 Python 代码使用了 Numpy 提供的svd()函数获得训练集的所有主成分,然后提取前两个 PC:
X_centered=X-X.mean(axis=0)U,s,V=np.linalg.svd(X_centered)c1=V.T[:,0]c2=V.T[:,1]
警告:PCA 假定数据集以原点为中心。正如我们将看到的,Scikit-Learn 的
PCA类负责为您的数据集中心化处理。但是,如果您自己实现 PCA(如前面的示例所示),或者如果您使用其他库,不要忘记首先要先对数据做中心化处理。
投影到d维空间
一旦确定了所有的主成分,你就可以通过将数据集投影到由前d个主成分构成的超平面上,从而将数据集的维数降至d维。选择这个超平面可以确保投影将保留尽可能多的方差。例如,在图 8-2 中,3D 数据集被投影到由前两个主成分定义的 2D 平面,保留了大部分数据集的方差。因此,2D 投影看起来非常像原始 3D 数据集。
为了将训练集投影到超平面上,可以简单地通过计算训练集矩阵X和Wd的点积,Wd定义为包含前d个主成分的矩阵(即由V^T的前d列组成的矩阵),如公式 8-2 所示。
公式 8-2 将训练集投影到d维空间
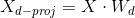
下面的 Python 代码将训练集投影到由前两个主成分定义的超平面上:
W2=V.T[:,:2]X2D=X_centered.dot(W2)
好了你已经知道这个东西了!你现在已经知道如何给任何一个数据集降维而又能尽可能的保留原数据集的方差了。
使用 Scikit-Learn
Scikit-Learn 的 PCA 类使用 SVD 分解来实现,就像我们之前做的那样。以下代码应用 PCA 将数据集的维度降至两维(请注意,它会自动处理数据的中心化):
from sklearn.decomposition import PCApca=PCA(n_components=2)X2D=pca.fit_transform(X)
将 PCA 转化器应用于数据集后,可以使用components_访问每一个主成分(注意,它返回以 PC 作为水平向量的矩阵,因此,如果我们想要获得第一个主成分则可以写成pca.components_.T[:,0])。
方差解释率(Explained Variance Ratio)
另一个非常有用的信息是每个主成分的方差解释率,可通过explained_variance_ratio_变量获得。它表示位于每个主成分轴上的数据集方差的比例。例如,让我们看一下图 8-2 中表示的三维数据集前两个分量的方差解释率:
>>> print(pca.explained_variance_ratio_)array([0.84248607, 0.14631839])
这表明,84.2% 的数据集方差位于第一轴,14.6% 的方差位于第二轴。第三轴的这一比例不到1.2%,因此可以认为它可能没有包含什么信息。
选择正确的维度
通常我们倾向于选择加起来到方差解释率能够达到足够占比(例如 95%)的维度的数量,而不是任意选择要降低到的维度数量。当然,除非您正在为数据可视化而降低维度 — 在这种情况下,您通常希望将维度降低到 2 或 3。
下面的代码在不降维的情况下进行 PCA,然后计算出保留训练集方差 95% 所需的最小维数:
pca=PCA()pac.fit(X)cumsum=np.cumsum(pca.explained_variance_ratio_)d=np.argmax(cumsum>=0.95)+1
你可以设置n_components = d并再次运行 PCA。但是,有一个更好的选择:不指定你想要保留的主成分个数,而是将n_components设置为 0.0 到 1.0 之间的浮点数,表明您希望保留的方差比率:
pca=PCA(n_components=0.95)X_reduced=pca.fit_transform(X)
另一种选择是画出方差解释率关于维数的函数(简单地绘制cumsum;参见图 8-8)。曲线中通常会有一个肘部,方差解释率停止快速增长。您可以将其视为数据集的真正的维度。在这种情况下,您可以看到将维度降低到大约100个维度不会失去太多的可解释方差。
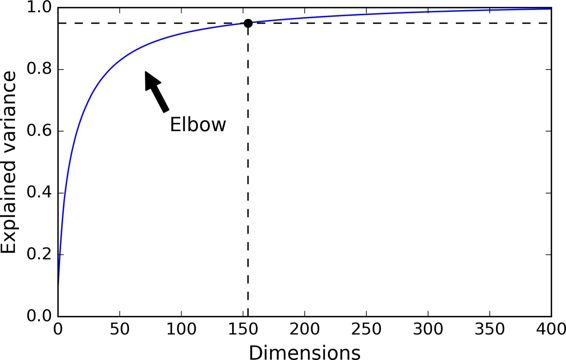
图 8-8 可解释方差关于维数的函数
PCA 压缩
显然,在降维之后,训练集占用的空间要少得多。例如,尝试将 PCA 应用于 MNIST 数据集,同时保留 95% 的方差。你应该发现每个实例只有 150 多个特征,而不是原来的 784 个特征。因此,尽管大部分方差都保留下来,但数据集现在还不到其原始大小的 20%!这是一个合理的压缩比率,您可以看到这可以如何极大地加快分类算法(如 SVM 分类器)的速度。
通过应用 PCA 投影的逆变换,也可以将缩小的数据集解压缩回 784 维。当然这并不会返回给你最原始的数据,因为投影丢失了一些信息(在5%的方差内),但它可能非常接近原始数据。原始数据和重构数据之间的均方距离(压缩然后解压缩)被称为重构误差(reconstruction error)。例如,下面的代码将 MNIST 数据集压缩到 154 维,然后使用inverse_transform()方法将其解压缩回 784 维。图 8-9 显示了原始训练集(左侧)的几位数字在压缩并解压缩后(右侧)的对应数字。您可以看到有轻微的图像质量降低,但数字仍然大部分完好无损。
pca=PCA(n_components=154)X_mnist_reduced=pca.fit_transform(X_mnist)X_mnist_recovered=pca.inverse_transform(X_mnist_reduced)

图 8-9 MNIST 保留 95 方差的压缩
逆变换的公式如公式 8-3 所示
公式 8-3 PCA逆变换,回退到原来的数据维度
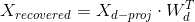
增量 PCA(Incremental PCA)
先前 PCA 实现的一个问题是它需要在内存中处理整个训练集以便 SVD 算法运行。幸运的是,我们已经开发了增量 PCA(IPCA)算法:您可以将训练集分批,并一次只对一个批量使用 IPCA 算法。这对大型训练集非常有用,并且可以在线应用 PCA(即在新实例到达时即时运行)。
下面的代码将 MNIST 数据集分成 100 个小批量(使用 NumPy 的array_split()函数),并将它们提供给 Scikit-Learn 的IncrementalPCA类,以将 MNIST 数据集的维度降低到 154 维(就像以前一样)。请注意,您必须对每个最小批次调用partial_fit()方法,而不是对整个训练集使用fit()方法:
from sklearn.decomposition import IncrementalPCAn_batches=100inc_pca=IncrementalPCA(n_components=154)for X_batch in np.array_spplit(X_mnist,n_batches):inc_pca.partial_fit(X_batch)X_mnist_reduced=inc_pca.transform(X_mnist)
或者,您可以使用 NumPy 的memmap类,它允许您操作存储在磁盘上二进制文件中的大型数组,就好像它完全在内存中;该类仅在需要时加载内存中所需的数据。由于增量 PCA 类在任何时间内仅使用数组的一小部分,因此内存使用量仍受到控制。这可以调用通常的fit()方法,如下面的代码所示:
X_mm=np.memmap(filename,dtype='float32',mode='readonly',shape=(m,n))batch_size=m//n_batchesinc_pca=IncrementalPCA(n_components=154,batch_size=batch_size)inc_pca.fit(X_mm)
随机 PCA(Randomized PCA)
Scikit-Learn 提供了另一种执行 PCA 的选择,称为随机 PCA。这是一种随机算法,可以快速找到前d个主成分的近似值。它的计算复杂度是O(m × d^2) + O(d^3),而不是O(m × n^2) + O(n^3),所以当d远小于n时,它比之前的算法快得多。
rnd_pca=PCA(n_components=154,svd_solver='randomized')X_reduced=rnd_pca.fit_transform(X_mnist)
